Inteligencia artificial predice la forma de las proteínas humanas y abre nueva era en la biología - El Mostrador
Científicos anunciaron en la revista Nature la disponibilidad de la mayor base de datos de proteínas que forman las estructuras de la vida, lo que "cambiará fundamentalmente la investigación en biología", según especialistas.
Piezas fundamentales de la vida, la estructura de cada proteína, que depende de los aminoácidos que la componen, define lo que hace y cómo lo hace, por lo que determinarla aportar información valiosa para entender los procesos biológicos, hacer avanzar diversos campos de investigar y podría servir para el desarrollo de fármacos.
Google DeepMind
La empresa DeepMind y el Laboratorio Europeo de Biología Molecular (EMBL) han empleado el sistema de inteligencia artificial AlphaFold para publicar la base de datos más completa y precisa de las predicciones de las estructuras de las proteínas humanas.
Cada célula de un organismo viviente ejecuta su función con la ayuda de proteínas que dan permanentemente instrucciones para mantener en buena salud la célula y combatir las infecciones. A diferencia del genoma –la secuencia de los genes que codifican la vida celular–, el proteoma humano cambia permanentemente en respuesta a instrucciones genéticas y estímulos exteriores.
La comprensión del funcionamiento de las proteínas, a través de la forma que adopten al interior de las células, es un verdadero desafío.
Solo se conoce el 17 % de los componentes del proteoma humano
Los científicos se han aplicado a determinar a través de experimentos su función precisa. Pero después de 50 años de investigación, solo se conoce el 17 % de los aminoácidos, o componentes del proteoma humano.
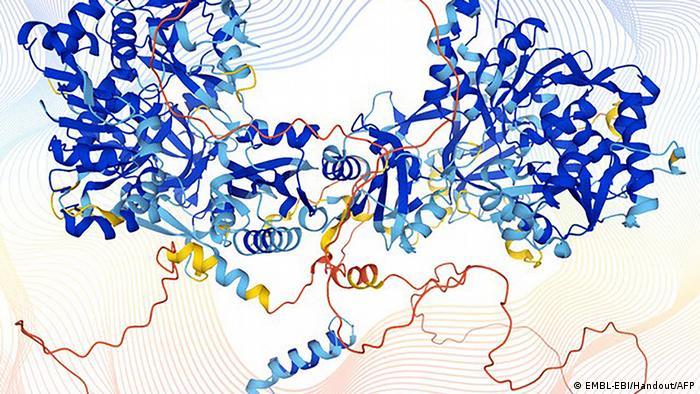
Los investigadores de Google DeepMind y del Laboratorio europeo de biología molecular (EMBL)revelaron el jueves una base de datos, de libre acceso, de 20.000 proteínas manifestadas por el genoma humano. A las que se agregan 350.000 proteínas de 20 organismos, como bacterias o ratones, utilizados para la investigación.
How to Boot from USB Drive to Install Windows, Ubuntu, or OSX on Surface Pro Devices - https://t.co/qnf4oR3QOR https://t.co/1OYs6mVidf
— alpinetworkcom Fri Apr 21 22:05:57 +0000 2017
AlphaFold: programa de aprendizaje automático
Esta base fue obtenida gracias a un programa de aprendizaje automático capaz de predecir con precisión la forma de una proteína a partir de su secuencia de aminoácidos.
El programa AlphaFold se entrenó con base a 170.000 estructuras conocidas de proteínas y luego predijo la forma del 58 % de todas las proteínas del proteoma humano, lo que más que duplicó el número de estructuras de proteínas humanas conocidas con precisión.
De ellos, la posición de un subconjunto del 35,7 % se predijo con un grado de confianza "muy alto", lo que supone el doble del número cubierto por las estructuras experimentales, explicó la revista.
Predicción de estructuras a gran escala
Los investigadores consideran que la predicción de estructuras a gran escala y con precisión se convertirá "en una herramienta importante que permitirá abordar nuevas cuestiones científicas desde una perspectiva estructural", y las predicciones de AlphaFold ayudarán a esclarecer aún más el papel de las proteínas.
"Creemos que esta es la contribución más significativa que ha hecho la inteligencia artificial al avance del conocimiento científico hasta la fecha, y es un gran ejemplo de los tipos de beneficios que la inteligencia artificial puede aportar a la sociedad", según el fundador de DeepMind, Demis Hassabis, firma británica que pertenece a Alphabet, matriz de Google.
El uso de la inteligencia artificial, con su capacidad de predecir computacionalmente la forma de una proteína a partir de su secuencia de aminoácidos, permite que no se tenga que determinar de forma experimental con el uso de técnicas laboriosas y a veces costosas.
Investigación sobre enfermedades genéticas
Las aplicaciones potenciales de estos datos van de la investigación sobre enfermedades genéticas a la ingeniería de cosechas resistentes a la sequía.
Según Paul Nurse, premio Nobel de medicina y director del Instituto Francis Crick, este avance es "un gran paso para la innovación en biología".
John McGeehan, director del Centro de innovación de Enzimas de la Universidad de Portsmouth, subrayó que "lo que tomaba meses y años en cumplirse fue realizado en un fin de semana por AlphaFold".
La capacidad de predecir con un programa informático la forma de una proteína a partir de su secuencia de ácidos aminados ya se está aplicando en algunos sectores de la investigación.
